See on Scoop.it – Med News
Ecco un esempio tutto italiano – dall’Università di Pavia – di conoscenze scientifiche trasferite alla vita quotidiana. Miconet, uno spin-off accademico fondato da 8 ricercatori provenienti dalla micologia, dalla botanica e dalla fisiologia, guarda con particolare attenzione alla nostra salute alimentare. Il background scientifico di questi ricercatori consente infatti di monitorare costantemente le proprietà funzionali e le caratteristiche nutritive di specifici prodotti che vengono poi proposti sul mercato: avremo quindi la possibilità di scegliere alcuni funghi dalle particolari proprietà officinali e vari alimenti derivati da essi.
See on www.miconet-italy.com
Biologia
12 novembre 2012
Miconet: i funghi dalla ricerca alla tavola
Posted by roberto insolia under Biologia, News, NutrizioneLascia un commento
21 ottobre 2012
Come l’Uomo si è adattato all’arsenico
Posted by roberto insolia under Biologia, GeneticaLascia un commento
See on Scoop.it – Med News
E’ noto come l’arsenico sia letale per quasi tutti gli organismi; tanto che la presunta scoperta nel 2010 di un batterio che addirittura conteneva nel proprio DNA questo elemento al posto del fosforo aveva destato enorme scalpore http://goo.gl/U7b7I Sono stati ora presentati i primi risultati ottenuti da alcuni ricercatori che da tempo studiano le popolazioni di quei villaggi sulle Ande argentine, dove l’acqua contiene alti livelli di arsenico. Da migliaia di anni queste popolazioni ingeriscono quantità potenzialmente tossiche di arsenico, senza apparentemente accusare alcun effetto: questo potrebbe essere legato a una specifica variante genetica del gene AS3MT, il principale responsabile del metabolismo cellulare dell’arsenico. Questa specifica variante è infatti molto più comune nella popolazione dei villaggi argentini con l’acqua ricca di arsenico, rispetto ad altre popolazioni rurali del Perù, dove invece questo elemento è pressoché assente nell’acqua. Se i dati saranno confermati, tutto questo rappresenterebbe uno dei pochi esempi – fin’ora noti nello scenario dell’evoluzione Umana – di adattamento alle tossine ambientali.
See on ehp.niehs.nih.gov
1 ottobre 2012
Open access: la lista nera delle riviste
Posted by roberto insolia under Biologia, Genetica, Medicina, NewsLascia un commento
See on Scoop.it – Med News
L’invezione dell’email ha contribuito enormemente ad accelerare la comunicazione; poi è arrivato lo spam che ha ridotto l’efficacia della posta elettronica. Allo stesso modo, prima si è sviluppato il mercato delle pubblicazioni “open access”, che certamente ha portato a una maggiore diffusione della conoscenza scientifica; poi sono nate anche delle riviste open access dal dubbio valore scientifico, che di fatto hanno abbassato la qualità globale delle pubblicazioni. Questo è in sintesi lo scenario dell’editoria tecnico scientifica – almeno in una parte di essa. Ecco quindi che Jeffrey Beall, bibliotecario alla University of Colorado, pubblica e aggiorna la lista delle riviste open access ritenute “scadenti”; e ricorda ad ogni ricercatore come sia sua precisa responsabilità decidere dove è meglio pubblicare i dati di una ricerca, così come auspica che gli articoli pubblicati sulle riviste dal dubbio valore siano esaminati con estrema cura, durante il processo di valutazione.
See on scholarlyoa.com
27 agosto 2012
Una notizia molto preoccupante è stata pubblicata, durante le recenti vacanze estive, sulla rivista scientifica Nature. Dopo uno studio in merito alle conseguenze delle radiazioni di Fukushima sulla popolazione locale di uccelli [ http://goo.gl/7Qufb ], un gruppo di ricercatori giapponesi ha indagato 144 esemplari adulti di piccole farfalle azzurre (Zizeeria maha); queste farfalle erano state catturate, a partire da due mesi dopo l’incidente nucleare.
Sono state studiate le prime generazioni “più vecchie” e poi quelle successive, i cui genitori erano adulti all’epoca dello scoppio dei reattori di Fukushima. Confrontando le alterazioni presenti in farfalle raccolte in luoghi diversi, si è visto che le zone con più alta quantità di radiazioni ambientali erano quelle che ospitavano farfalle co ali molto più piccole e occhi sviluppati in modo irregolare. In particolare, queste anomalie aumentavano passando dalle prime generazioni a quelle più giovani, suggerendo l’esistenza di alterazioni genetiche che venivano ereditate da una generazione all’altra. Questo studio è importante, soprattutto per le implicazioni che potrebbe avere per le altre specie, Uomo compreso ovviamente, che vivevano e vivono tuttora nella zona di Fukushima.
See on www.nature.com
31 luglio 2012
Zanzare: il virus dell’encefalite giapponese in Italia?
Posted by roberto insolia under Biologia, NeurologiaLascia un commento
Nell’estate del 2010 e del 2011, un gruppo di ricercatori italiani ha campionato le zanzare che infestavano 8 località diverse nella zona fra Bologna e Modena, con lo scopo di studiare l’infezione dei flavivirus a RNA [ http://goo.gl/CUCiF ], veicolati da questi insetti. Dai campioni raccolti sono stati sequenziati tre diversi genomi: quello di un virus specifico degli insetti, quello del già descritto virus Usutu e, cosa più inaspettata, una sequenza genomica che ricalca in parte quella del virus dell’encefalite giapponese (JEV). Mentre i primi due sono già stati identificati in Italia e in altri Paesi europei, il JEV non era mai stato isolato dalle zanzare dell’Europa; è questo un flavivirus, trasmesso dalle zanzare ed endemico in Asia, dove è la principale causa di encefalite.
In realtà, gli stessi autori dello studio sono molto cauti su questo dato. La sequenza genomica, che corrisponde a quella identificata in alcuni ceppi di JEV isolati in pipistrelli cinesi, è infatti molto breve; inoltre è noto che flavivirus diversi presentano comunque alte omologie nel loro genoma. La sequenza individuata potrebbe non essere quella del JEV, ma di un altro flavivirus non ancora identificato. Inoltre, non è stato possibile amplificare zone più ampie del genoma né isolare in modo diretto il virus dell’encefalite giapponese nelle zanzare emiliane. Dal punto di vista epidemiologico, va detto che in Italia il sistema di sorveglianza delle malattie infettive non ha ancora registrato un caso di encefalite giapponese. Certo è che, come dichiara lo stesso dipartimento di Malattie Infettive, Parassitarie e Immunomediate dell’Istituto Superiore di Sanità [ http://goo.gl/yleAB ], bisogna essere vigili, sia per approfondire questo primo riscontro scientifico che per essere pronti ad applicare eventuali misure di intervento sanitario.
See on www.eurosurveillance.org
30 luglio 2012
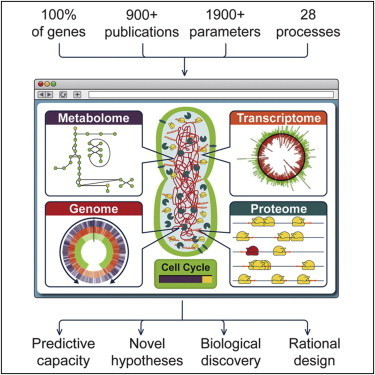
Nel 2010 il primo passo era stato quello di creare un cromosoma artificiale – cioè sintetizzato in modo chimico – all’interno di cellule che erano state in grado di svilupparsi normalmente [ http://goo.gl/pxw1a ]. Ora si è andati oltre, o meglio si è affrontato l’aspetto della “vita artificiale” da un punto di vista diverso.
Lo stesso gruppo di ricercatori impegnato nel 2010 ha infatti seguito il ciclo vitale di un batterio, simulandolo per la prima volta attraverso un computer. Tutto quello che avviene routinariamente in una cellula – cioè la sintesi di DNA e RNA, la traduzione e poi il funzionamento delle varie proteine, fino alla divisione della cellula stessa – sono stati convertiti in algoritmi di calcolo. La vita quindi di Mycoplasma genitalium, uno dei batteri più piccoli attualmente conosciuti, il cui DNA porta solamente 525 geni, è stata simulata attraverso l’impiego di 128 computer collegati fra loro.
“Attualmente, la duplicazione del batterio può essere simulata in 10 ore, producendo circa mezzo gigabyte di dati”, dichiara Markus Covert , il bioingeniere responsabile della ricerca. Immaginiamoci quanti computer e quale mole di dati serviranno per simulare l’attività dei 4.228 geni di Escherichia coli, il batterio più frequentemente impiegato in laboratorio. Certo è che l’avere a disposizione il modello virtuale di una cellula potrà velocizzare la ricerca sul ruolo dei singoli geni, sia in una cellula sana che in una malata, quale può essere quella tumorale.
See on www.cell.com
17 luglio 2012
Non avevamo parlato nel 2010 della scoperta di un batterio che “sembrava” avere come costituente fondamentale del proprio DNA l’arsenico, al posto del fosforo che è invece presente in tutti gli organismi viventi [ http://alturl.com/znig7 ]. Era una ricerca che vedeva coinvolta la NASA e che, se confermata, avrebbe avuto delle implicazioni biologiche molto importanti: l’arsenico è infatti letale per quasi tutti gli organismi. Avevamo invece visto, in materia di invecchiamento nell’Uomo, come un modello biostatistico fosse sì riuscito a individuare dei marcatori genetici legati all’invecchiamento, ma poi fosse anche stato necessario ritrattare, almeno in parte, tutti quei dati [ http://alturl.com/bzhpv ].
Comunque, il presunto batterio all’arsenico aveva sollevato molte riserve all’interno della comunità scientifica, tanto che Felisa Wolfe-Simon, primo autore della pubblicazione apparsa su Science, aveva a un certo punto smesso di rispondere ai quesiti e alle richieste di approfondimenti da parte dei colleghi ricercatori. Ora, sempre Science presenta due ricerche indipendenti che dimostrano come quel batterio in realtà debba comunque usare un po’ di fosforo per sopravvivere: il punto è che non è in grado di vivere solo (!) in presenza di arsenico, ma più semplicemente riesce a evitarne gli effetti tossici [ http://alturl.com/t8boh http://alturl.com/gkb2n ].
Quindi, se le basi genetiche dell’invecchiamento umano sono qualcosa di molto complesso, anche il batterio “all’arsenico” è da studiare con estremo rigore scientifico. Tanto che Science stessa ci ricorda come il processo scientifico, e quindi la sua comunicazione, siano in grado di auto-correggersi, dal momento che i ricercatori stessi cercano di verificare i risultati precedentemente ottenuti dai colleghi. Perciò, “la rivista Science pubblica ora ulteriori informazioni su un batterio particolarmente resistente, in grado di aiutare la comprensione dei meccanismi di resistenza all’arsenico”.
See on www.sciencemag.org
20 giugno 2012
Censimento batterico nel nostro organismo
Posted by roberto insolia under Biologia, MedicinaLascia un commento
Oltre 10.000 specie diverse. E’ questo il risultato del censimento microbico di oltre 5.000 campioni biologici – relativi a 18 differenti campioni di tessuto raccolti in 242 (113 donne) americani sani – frutto dello Human Microbiome Project (HMP) Consortium [ http://alturl.com/gs772 ]; è questa una collaborazione fra circa 80 centri di ricerca che, con le più avanzate tecnologie molecolari nell’arco di cinque anni, ha sequenziato il DNA dei batteri che comunemente sono presenti nel nostro organismo.
Il primo inaspettato risultato è proprio quello del loro enorme numero: per intenderci, per ogni cellula umana ci sono 10 cellule batteriche. Oltre a questo, sorprende anche il fatto che non sembra esserci qualcosa di completamente condiviso fra tutti gli individui: ognuno di noi ha infatti il proprio carico microbico, in buona parte diverso da quello delle altre persone. Come se ogni singolo individuo fosse una sorta di ecosistema microbico, con la propria popolazione e con i propri equilibri biologici all’interno di essa. Poi, c’è stata la sfida bioinformatica: di fatto sono stati gestiti i dati per oltre 8 milioni di sequenze codificanti di DNA, cioè proteine – circa 360 volte il carico dei dati relativi all’organismo umano. E nel futuro bisognerà sviluppare gli strumenti di analisi più idonei per capire al meglio la funzione di tutte queste molecole.
Non ultimo, c’è il significato scientifico della cosa: a 10 anni dal completamento dello Human Genome Project [ http://alturl.com/ke2mb ], ecco che i primi risultati dello Human Microbiome Project rappresentano il punto di partenza per approfonditi studi sull’interazione fra l’Uomo e i batteri. Perché se solitamente questa coesistenza nel nostro organismo è pacifica, nel corso della vita il nostro sistema immunitario si trova anche a fronteggiare delle infezioni: sarà quindi importante comprendere come alcuni agenti patogeni piuttosto che altri potranno nel futuro minacciare la nostra salute. A dimostrazione anche della significatività di questo progetto sta il fatto che i risultati scientifici della ricerca sono stati presentati in diverse pubblicazioni, sulle principali riviste scientifiche. E in realtà, tutto questo ci porta anche a pensare come il nostro corpo non sia poi proprio tutto “nostro”.
See on www.nature.com
8 giugno 2012
L’evoluzione dell’Uomo mediata dalle infezioni
Posted by roberto insolia under BiologiaLascia un commento
E’ noto come la famiglia delle immunoglobuline comprenda alcune proteine di membrana, dette recettori, che possono legare l’acido sialico [ http://alturl.com/jxkt3 ], uno dei principali componenti della parete batterica; alcuni di questi recettori, sono quindi coinvolti nei processi infettivi, mediando l’ingresso degli agenti patogeni nell’organismo dell’ospite.
Circa 150.000 anni fa, la popolazione dei nostri antenati in Africa è stata decimata, probabilmente a seguito di grosse infezioni che sembravano avere la meglio sulle difese immunitarie degli ominidi di allora. Tuttavia, proprio in quell’età sono comparsi anche i nostri “più vicini” antenati – fra cui l’uomo di Neanderthal – dimostrando come in realtà fosse stato possibile combattere quelle infezioni. Ma cosa è biologicamente successo allora?
Un gruppo di antropologi e biologi molecolari ha ipotizzato che in una singola, ristretta popolazione di ominidi sia insorta una mutazione genetica che ha conferito una spiccata resistenza ai flagelli infettivi di quell’epoca: in particolare, potrebbe essere stata eliminata la molecola che consentiva la trasmissione degli agenti infettivi da un individuo all’altro. Infatti, è stato dimostrato che proprio i geni codificanti quei recettori dell’acido sialico sono praticamente assenti o inattivi nel nostro “moderno” genoma, mentre continuano a svolgere la loro funzione di mediatori dell’infezione nel genoma degli scimpanzé, i nostri cugini evolutivamente più vicini. Il fatto che quei geni, bersaglio dei patogeni, siano stati selettivamente spenti nel DNA di alcuni nostri antenati, potrebbe avere rappresentato la strategia evolutiva vincente per non estinguersi.
“Ecco la dimostrazione che il processo dell’evoluzione, attraverso il quale nuove specie possono comparire da altre già esistenti, è indirizzato da vari e differenti stimoli ambientali e biologici, fra cui vi sono certamente gli agenti infettivi”, chiarisce Ajit Varki, coordinatore dello studio.
See on www.pnas.org
7 giugno 2012
Perché la probabilità di sviluppare un tumore al seno aumenta con il progredire dell’età anagrafica della donna? La risposta a questo quesito sarebbe un passo avanti nella comprensione dei meccanismi biologici alla base della crescita tumorale e aiuterebbe lo sviluppo di terapie, non solo di cura nel caso di comparsa del tumore, ma anche di prevenzione.
Attingendo anche a numerosi campioni di tessuto congelato negli ultimi 30 anni, i ricercatori coordinati dal biologo Mark LaBarge del Lawrence Berkeley National Laboratory hanno coltivato in laboratorio le cellule epiteliali del tessuto mammario, prelevate da donne con età compresa fra i 16 e 91 anni. Creata questa banca di cellule, sono stati poi condotti studi di tipo cellulare, molecolare e biochimico, seguendo le cellule stesse nel corso della loro vita; di fatto quindi, vedendole invecchiare. In questo modo è stato dimostrato che durante l’invecchiamento aumenta il numero di specifiche cellule “multipotenti”, cioè quelle le cui caratteristiche sono tali da potersi trasformare in cellule tumorali. Diminuisce invece la quota delle cellule, che solitamente stanno accanto a quelle che producono il latte, e che sono ritenute dei soppressori della crescita tumorale. Il complesso rimaneggiamento nella popolazione delle cellule che invecchiano potrebbe quindi spiegare la più frequente comparsa di tumori con l’avanzamento dell’età.
Questa ricerca è importante, sia perché il 75% delle nuove diagnosi di tumore al seno si registra nelle donne oltre i 50 anni di età, sia per la tecnica impiegata nello studio delle cellule stesse. Non è infatti semplice indagare l’invecchiamento nelle cellule umane, tanto che solitamente si usano particolari “modelli” – quali il lievito, il lombrico, il topolino – i cui parametri cellulari sono più facilmente controllabili e misurabili in laboratorio. La tecnica di coltura cellulare messa a punto ora potrebbe addirittura essere applicata ad altri tessuti, per studiare in modo più generale le problematiche legate all’invecchiamento cellulare.
See on cancerres.aacrjournals.org